이 연구에서는 RAPD와 ISSR표식자들을 리용하여 소나무종들의 63개 생식질들의 유전적류연관계를 분석하였다.
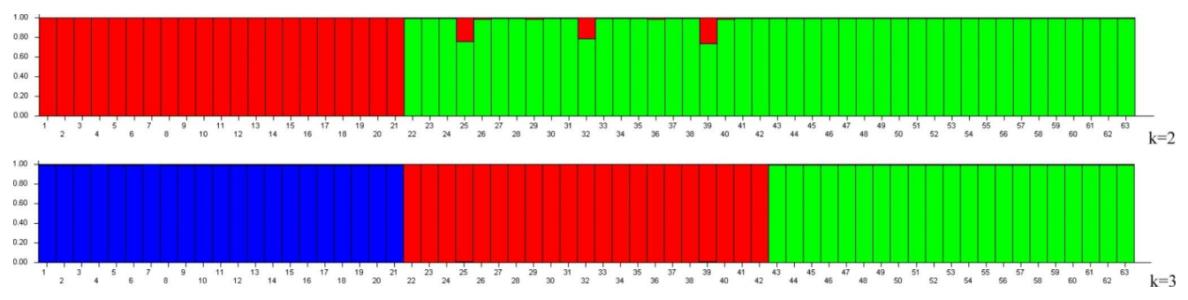
2개의 종과 품종집단들은 0.53-0.72의 높은 FST값을 나타내는 명백한 유전적차이를 보여주었다. PCA 및 UPGMA무리분석은 시료들을 종에 따라 기본무리들로 구분하였다.
먼저 소나무와 곰솔이 구분되고 다음 《금야흑송》으로 구분되였다. 바예시안구조분석은 K=3에서 《금야흑송》의 구분을 강조하였다. 《금야흑송》품종이 높은 근교를 나타냈음에도 그것은 곰솔에 비해 더 높은 관찰 및 유효대립유전자수와 다형을 가진다는것이 확인되였다.
이러한 결과들은 《금야흑송》의 품종적지위를 확인하며 또한 그의 기타 표현형적특징들이 독특한 유전적배경에 기인될수 있다는것을 시사해준다. 이러한 개체들의 자연섞붙임의 동정과 지문작성은 곰솔과 련관된 종들의 육종에서 재료를 효과적으로 선택하는데서 중요하다.
연구결과는 잡지 《Biologia》에 《Genetic relationship between Pinus species in the Democratic People's Republic of Korea》(https://doi.org/10.1007/s11756-023-01426-2)의 제목으로 출판되였다.